Molekulare Mikrobiologie
MOLEKULARe MIKROBIOLOGIE
Sepsis und andere schwere Infektionen werden hauptsächlich durch Bakterien oder Pilze verursacht. Die frühzeitige Identifizierung des verursachenden Erregers ist entscheidend für eine wirksame und erfolgreiche Behandlung mit Antibiotika, Antimykotika, unterstützenden Medikamenten und für begleitende Therapien. Die molekularen Erregertests von Cube Dx unterstützen Mikrobiologen und Spezialisten für Infektionskrankheiten bei der frühzeitigen Erkennung von Sepsis verursachenden Erregern in Vollblut und anderen Probenmatrices.
Besonders wichtig ist dies für Mikroorganismen die durch eine antimikrobielle Behandlung geschwächt wurden oder die in mikrobiologischen Nährmedien nur langsames Wachstum zeigen. Der molekulare Test von Cube Dx ist aufgrund seines breiten mikrobiellen Spektrums ideal dafür geeignet Erreger schwerer Infektionen wie einer Sepsis nachzuweisen.
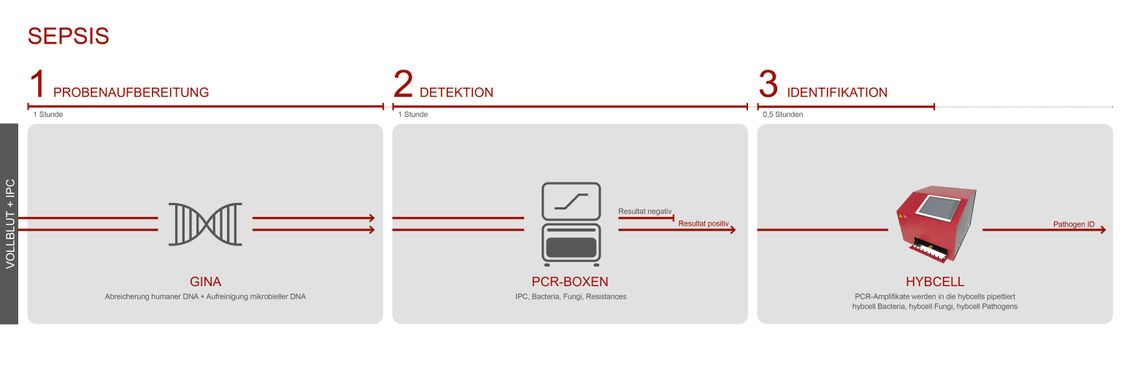
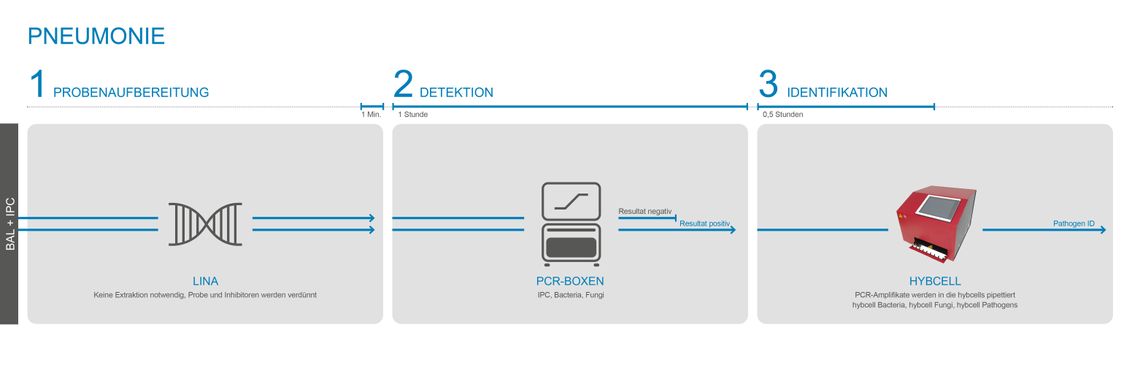
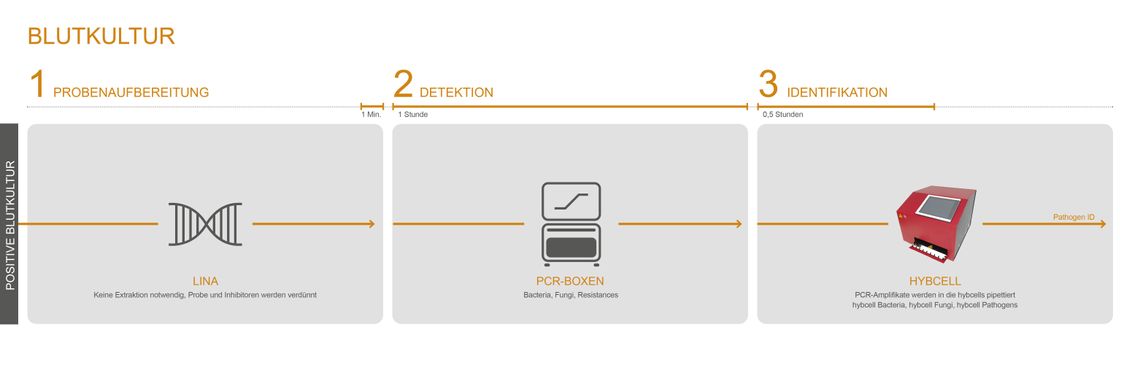
Der Test umfasst drei Prozesse: die Probenvorbereitung und DNA-Aufreinigung, pan-bakterielle bzw. pan-fungale PCR und die Identifikation des Erregers mit der hybcell.
Das PCR-Screening für Bakterien (16S) und Pilze (28S) und die anschließende Identifikation auf der hybcell werden als compact sequencing bezeichnet. Die Testkomponenten (PCR-Mastermixe und hybcells) und der Ablauf sind für alle untenstehenden Zweckbestimmungen grundsätzlich gleich. Die Probenvorbereitung unterscheidet sich jedoch stark zwischen Vollblutproben und BAL / positiven Blutkulturen für den Nachweis einer Pneumonie oder anderen Infektionen.
Während bei der Analyse von Vollblut die humane DNA mit dem GINA-Kit entfernt wird ist bei BAL oder positiven Blutkulturen keine DNA-Extraktion erforderlich. Sattdessen wird der Modulationspuffer LINA verwendet.
87 Mikroorganismen / Resistenzgene - in 3 Stunden
SEPSISDIAGNOSE - OHNE KULTIVIERUNG / AUS BLUT
Cube Dx unterstützt die behandelnden Ärzte bei Verdacht auf eine Sepsis indem der Test einen sehr frühen Hinweis auf Sepsis verursachende Mikroorganismen direkt aus Vollblut liefert. Der Test verkürzt die Zeit bis zum Ergebnis auf nur 3 Stunden!
Unsere Multiplex-Technologie ermöglicht die gleichzeitige Identifizierung von 87 Targets (Mikroorganismen und Resistenzgene).
Als Probe dient 0,5mL frisches EDTA-Blut. Zur Validierung eines negativen Ergebnisses kann optional eine interne Prozesskontrolle (IPC) in die Probe gegeben werden.
Während des GINA-Prozesses wird die Mehrzahl der menschlichen Zellen abgetrennt, bevor die DNA gereinigt wird.
Die angereicherte mikrobielle DNA wird mit den PCR-Mastermixen amplifiziert: der Anwender wählt je nach klinischer Situation alle oder einzelne aus den zur Verfügung stehenden PCR-Mixen aus (pan-bakterielle, pan-fungale PCR und eine Multiplex-PCR auf verschiedene Resistenzgene).
Positive Proben werden in die hybcell-Kartuschen überführt und die Mikroorganismen und Resistenzgene durch das compact sequencing identifiziert. Die Ergebnisse werden automatisch in einem kompakten Bericht zusammengefasst.
Eigenschaften / Vorteile:
-
Ergebnisse in nur 3 Stunden!
-
Bis zu 98% Ausbeute an mikrobieller DNA
-
Nachweisgrenze (Vollblut): 20 KBE/mL
-
Klinische Sensitivität: 74% und Spezifität: 98%
-
Frühe und umfangreiche Ergebnisse aus nur 0,5 ml Vollblut
-
Sensible und parallele Identifizierung von Bakterien, Pilzen und Resistenzgenen
-
Identifizierung mehrerer Erreger/Mischinfektionen
-
Einfache Handhabung, optimierter Workflow mit 24/7-Verfügbarkeit und sofortigen Ergebnissen
31 Mikroorganismen - in 2 Stunden
DIAGNOSE EINER LUNGENENTZÜNDUNG - OHNE EXTRAKTION / AUS BAL
Mit den hochempfindlichen PCR-Mastermixen von Cube Dx kann BAL direkt getestet werden - ohne dass eine DNA-Extraktion erforderlich ist. Ein einfacher Modulationspuffer - LINA - wird zur Modulation und Homogenisierung der BAL-Probe verwendet. Durch die Verwendung des extraktionsfreien Protokolls verkürzt sich die Zeit bis zum Ergebnis auf nur 2 Stunden.
Der LINA-Modulationspuffer wird für Proben mit einer relativ hohen Konzentration von Erregern, z. B. BAL, verwendet. Nur wenige µl BAL werden in das gebrauchsfertige LINA-Reaktionsgefäß gegeben und die Lösung kurz gemischt. Die Mischung wird anschließende direkt für die PCR verwendet. Dabei werden die gleichen Produkte (PCR und hybcell) wie für die Testung der Sepsiserreger verwendet. Eine Softwareoption beschränkt die Ausgabe auf das CE-IVD-zertifizierte Panel für Pneumonie.
Der LINA-Transfer- und Modulationspuffer verkürzt die Zeit für die molekulare Identifizierung auf 2 Stunden, weil der DNA-Extraktionsprozess entfällt.
Eigenschaften/Vorteile
-
Keine DNA-Extraktion
-
Identifizierung von Krankheitserregern in weniger als 2 Stunden
-
Einfache Handhabung, wenige manuelle Arbeitsschritte
-
Sensible und parallele Identifizierung von Bakterien und Pilzen
-
Identifizierung mehrerer Erreger/Mischinfektionen
100 Mikroorganismen / Resistenzgene - in 2 Stunden
MIKROBIELLE IDENTIFIZIERUNG - DIREKT AUS POSITIVER BLUTKULTUR
Cube Dx bietet eine schnelle DNA-basierte Identifizierung von Mikroorganismen die in Blutkulturen gewachsen sind. Es ist keine weitere Reinkultur erforderlich und selbst langsam wachsende Mikroorganismen können parallel zum dominierenden Erreger identifiziert werden. Der Test kann beispielsweise als Backup-System zu einer MALDI-TOF basierten Identifizierung etabliert und verwendet werden.
Der LINA-Puffer bietet einen einfachen, schnellen und effizienten Weg zur Identifizierung krankheitsverursachender Erreger mit nur 1 Minute Zeit für die Handhabung. Wenige µl einer positiven Blutkultur können direkt in den LINA-Puffer gegeben werden ohne dass eine Reinkultur erforderlich ist. Das Gemisch wird in die PCR überführt und die Identifizierung erfolgt mit der hybcell - das Ergebnis wird in 2 Stunden geliefert.
Eigenschaften/Vorteile
-
Bakterien: 56 Spezies + 11 Genera
-
Fungi: 19 Spezies + 5 Genera
-
9 Resistenzgene
-
Nachweis von Mischinfektionen
-
Keine DNA-Extraktion
-
Identifizierung von Krankheitserregern in weniger als 2 Stunden
-
Einfache Handhabung, wenige manuelle Arbeitsschritte
-
Identifizierung von Mehrfacherregern/Mischinfektionen
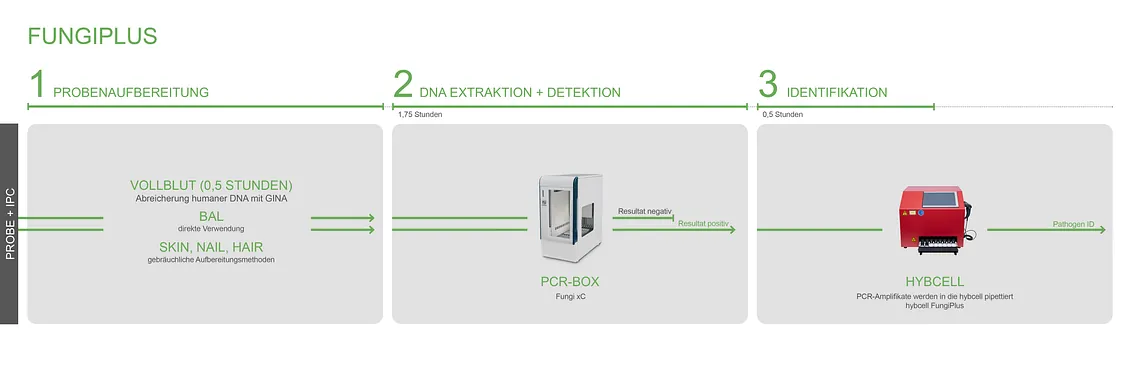
16 Pilzgenera / 50 Pilzspezies - in 2,5 Stunden
FÜR FoRSCHUNGSZWECKE - BREITER MOLEKULARER PILZTEST
Cube Dx bietet eine schnelle und umfangreiche DNA-basierte Identifizierung von Pilzen aus unterschiedlichen Proben für Forschungszwecke. Durch die Verwendung unsere GINA-Probenaufbereitung kann auch die Gesamtzeit zum Resultat für "schwierige" Proben wie Haut- oder Nagelschuppen drastisch verkürzt werden.
Als Probe kann beispielsweise 0,5mL frisches EDTA-Blut dienen, die mit der GINA Probenaufbereitung vorbereitet wird. Es können aber auch jegliche andere Proben - von BAL über Urin, CSF, Gewebe bis hin zu Haut, Nägel oder Haare - verwendet werden. Als Eingangsmaterial des Tests dient demnach die im Eluat enthaltende Proben-DNA.
Die angereicherte mikrobielle DNA wird mit den PCR-Mastermix amplifiziert und positive Proben werden in die hybcell-Kartuschen überführt und die Pilze durch das compact sequencing identifiziert. Die Ergebnisse werden automatisch in einem kompakten Bericht zusammengefasst.
Eigenschaften/Vorteile
-
16 Pilzgenera
-
50 Pilzspezies
-
66 Pilztargets in 2,5 Stunden
-
Testung unterschiedlichster Proben
-
Automatisierter Ablauf mit wenigen manuellen Arbeitsschritte
-
Identifizierung von Mehrfacherregern/Mischinfektionen
Molekulare Mikrobiologie – Literatur
VERFÜGBARE LITERATUR
Verfügbare Literatur:
- 2016: Sensitive Molecular Identification of Pathogens causing Implant and Tissue Infections (ITI)
- 2020: Molecular pathogen identification and resistance gene detection from positive blood cultures
- 2020: Presence and distribution of fungal species and dermatophytes in nail and skin samples
- 2021: SMARTDIAGNOS - next generation molecular sepsis diagnosis technology for whole blood samples
- 2022: Extraction-free Molecular Identification of Bacteria/Fungi Directly from BAL Samples
- 2023: Comparison of two molecular assays and MALDI-TOF MS Sepsityper for the rapid identification of pathogens from positive blood cultures
- 2023: Detection of bacterial pneumonia pathogens in patients with COVID-19
- 2023: Diagnostic value of blood culture independent molecular tests for the diagnosis of bloodstream infections in neutropenic patients with fever